欢迎回来,十月下系列因为一些个人的事情有些摸鱼,十一月作为新的开始,希望Flag能立住。
11月的歌,祝贺EDG!
《新生》这首歌原本是LOL十周年的歌,但感觉很符合大家在世界赛对EDG的看法,歌本身很好听,很有古风的感觉,前面的女声也很空灵。
[title]2021年11月2日[/title]
Deep Learning-Based Prediction of Drug-Induced Cardiotoxicity
JCIM 2019 广州中医药大学的文章,通过药物的分子式预测其是否为特定浓度下的hERG阻滞剂。文章的主要贡献是
- 自己收集了一个很大的药物数据库作为模型的输入
- 提出了一套处理药物分子式的pipeline,当然不知道其他文章是不是也这么做的,利用一些最小化能量等我看不懂的操作对输入做了预处理
- 训了一个0.9ROC的模型,并在癌症药物上做了验证,和一以后的文献报到相互印证。
放上文章的Pipeline,不太需要解释应该都可以看懂:文章里提到了MLP的效果比图卷积要好用,不知道是不是图卷积的使用有什么问题导致的。
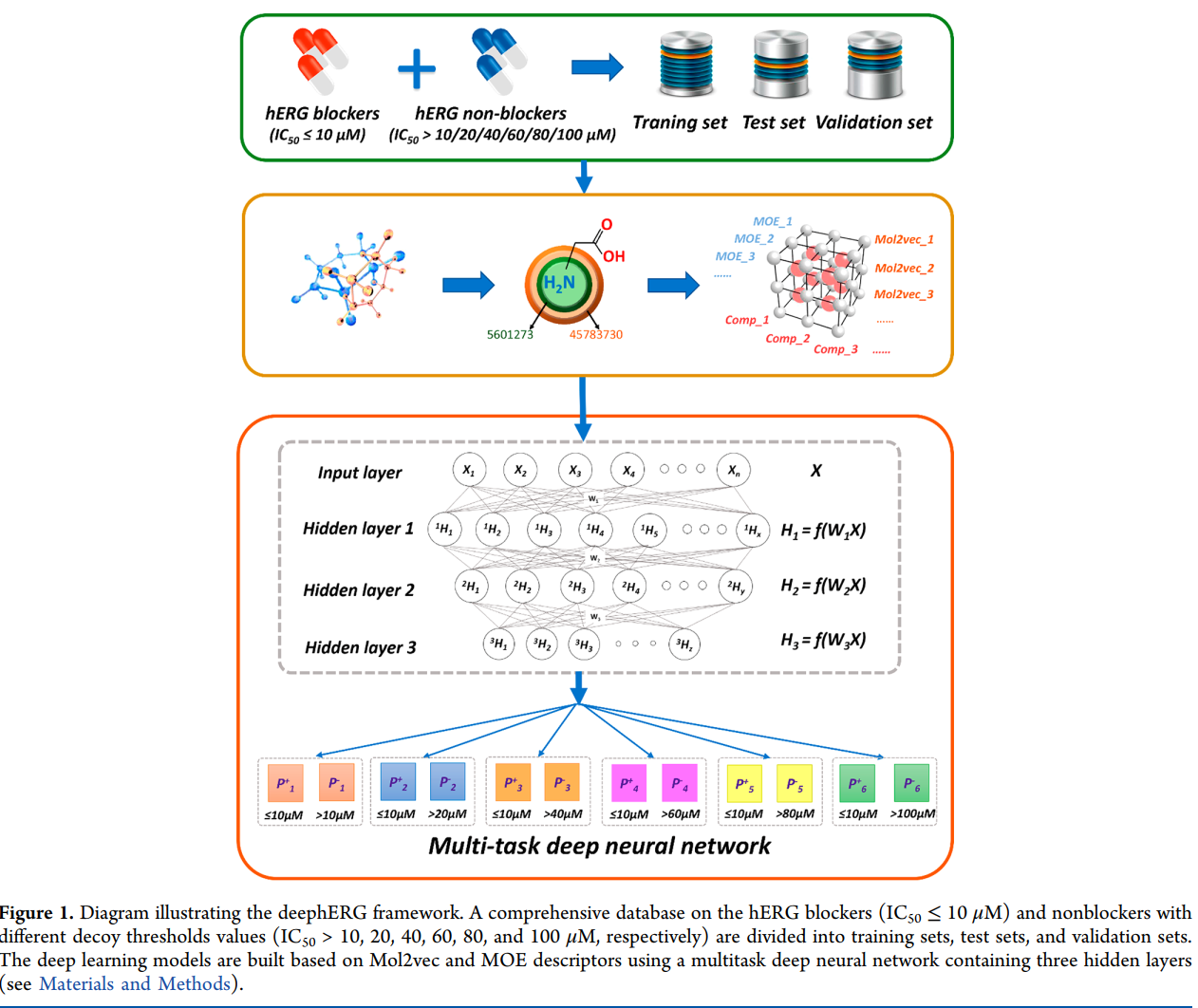
[title]2021年11月3日[/title]
Contrastive learning of global and local features for medical image segmentation with limited annotations
组会 NIPS 2020 一篇在心脏MRI图像上做 Contrastative loss的文章,因为MRI是三维的图片,所以作者提出了“四横四纵”的对比学习策略,第一个loss是在Z轴上切4层,然后将不同人对应的同一层认为是相似的,而不在同一层(份)的认为不同,这样构建一个损失。第二个loss是对于每一张MRI切片分成4份,将在同一层并且在同一份的图片对认为是相似的,而把在同一层但是不在同一份的认为是不一样的。
通过这样的预训练方式,作者最终就训练好了一个Unet,并且效果都比其他的要好。
第一个loss:
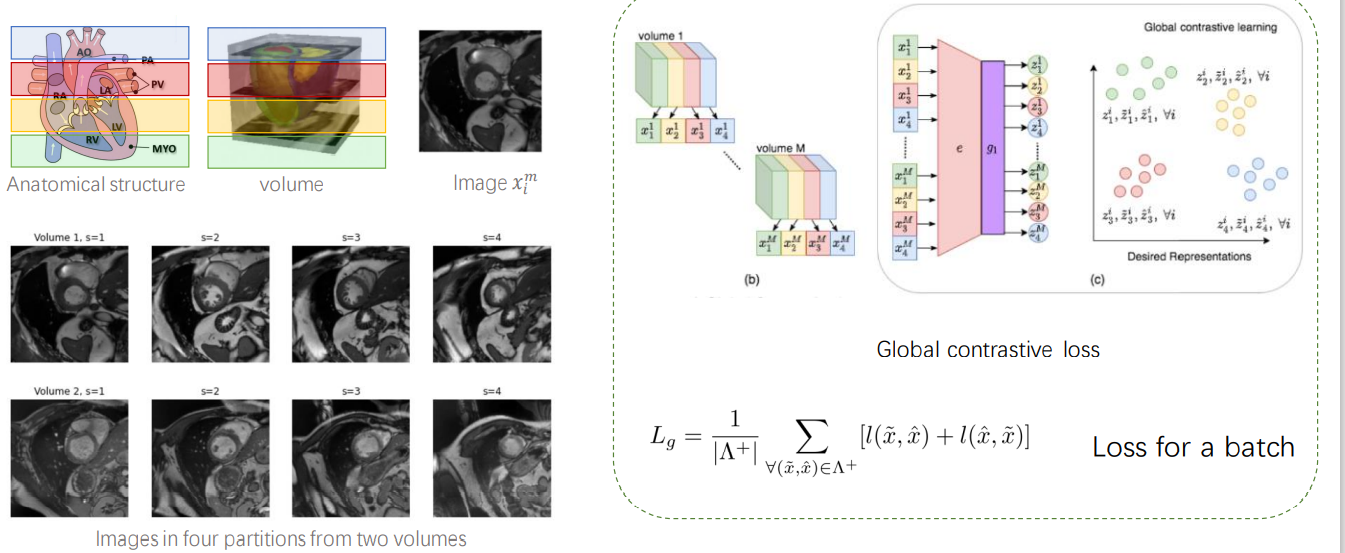
第二个loss:

[title]2021年11月04日[/title]
Ordinal Regression Methods: Survey and Experimental Study
IEEE TRANSACTIONS ON KNOWLEDGE AND DATA ENGINEERING 2016 今天读到了利用SVM实现序列回归问题的解法,思路很简单,就是在规定权重向量的情况下,设置不同的偏移量b,满足$b_1<b_2<...<b_{Q-1}$来将样本进行划分。但是我们知道,在仅进行多分类SVM时,无法保证偏移量的大小可以根据样本的标签大小进行排序,所以W. Chu提出了一系列利用所有数据对每一个分类面进行约束的方法,可以保证上面的偏移量约束。方法的公式如下所示,$N_q$ 是类别$C_q$的样本个数,其中样本为$x_qi$ ,那么公式为:
$$
\min _{\mathbf{w}, \mathbf{b}, \xi, \xi^{*}} \frac{1}{2}\|\mathbf{w}\|_{2}^{2}+C \sum_{q=1}^{Q-1}\left(\sum_{j=1}^{q} \sum_{i=1}^{N_{q}} \xi_{j i}^{q}+\sum_{j=q+1}^{Q} \sum_{i=1}^{N_{q}} \xi_{j i}^{* q}\right)
$$
约束条件为
$$
\begin{array}{ll}
\mathbf{w} \cdot \mathbf{x}_{i}^{j}-b_{q} \leq-1+\xi_{j i}^{q}, & \xi_{j i}^{q} \geq 0, \quad j \in\{1, \ldots, q\} \\
\mathbf{w} \cdot \mathbf{x}_{i}^{j}-b_{q} \geq+1-\xi_{j i}^{* q}, & \xi_{j i}^{* q} \geq 0, \quad j \in\{q+1, \ldots, Q\}
\end{array}
$$
可以看到这个和常见的C-SVM非常相似,就是通过控制$b$的位置,使得分类面左右两边的数据都恰好属于大于q和小于q这两类,本质上还是通过实现多个二分类来实现序列回归。
[title]2021年11月05日[/title]
QTNet: Predicting Drug-Induced QT prolongation with Deep Neural Networks
medRxiv 2021 一篇很短的letter,作者是纽约大学的医学博士们,在4628个病人上,拿到了服药前6个月内的心电图数据,以及相应的已知风险因素,去预测吃药后QT区间的变化情况(QTc>500ms或者$\Delta$QTC>60ms)。在4628个病人中,发生了QTc延长的有1030个病人。在药物的选择上,作者选取了“Fluconazole, Hydroxychloroquine, Amiodarone, Donepezil, Escitalopram, Levofloxacin, Citalopram, Erythromycin, Ciprofloxacin, Haloperidol, and Dronedarone”,这几类药物,也就是“氟康唑、羟氯喹、胺碘酮、多奈哌齐、艾司西酞普兰、左氧氟沙星、西酞普兰、红霉素、环丙沙星、氟哌啶醇、卓尼达隆”主要是抗心律药物和抗生素。作者没有详细说是如何确定观测QTc区间的。
作者比较了5个模型:1,RISQ-PATH 模型 2,对照QTC模型,没太懂这个对照QTc是哪里来的 3,ECG+CF(危险因素)4,ECG 5,CF。五个模型里效果最好的是ECG+CF,也就是QTnet,达到了0.802的ROC,而其他的达到了0.77左右,感觉提升不是很明显,我觉得是因为模型里没有考虑到药物的区别导致的,但作者也没有汇报药物的个数,不知道会不会是因为数据不支持。
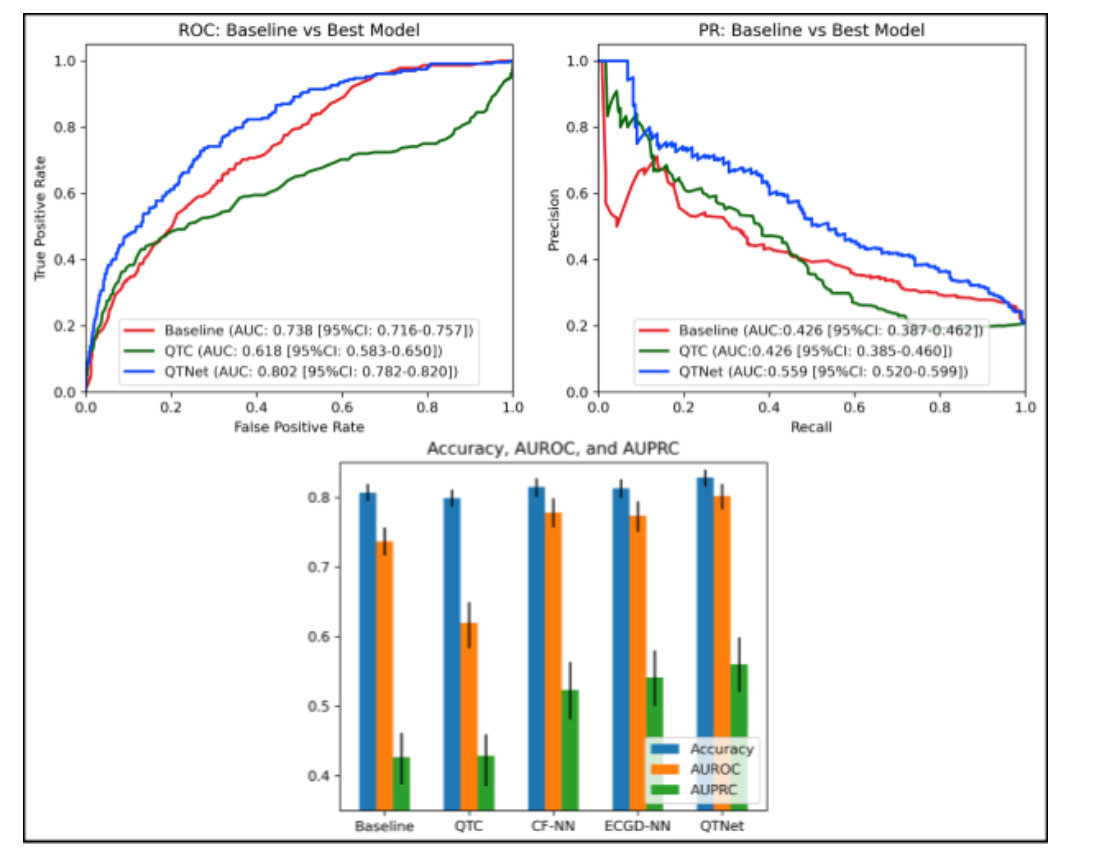
[title]2021年11月06日[/title]
A smart algorithm for the prevention and risk management of QTc prolongation based on the optimized RISQ-PATH model: The RISQ-PATH-model for QTc prolongation
BJCP 2018 在超大队列上对之前提出的RISQ-PATH模型进行定量研究的工作,这里的定量就是确定参数的权重。一共来自于17个医院的60,208个患者参与了研究,每个病人都有他正常时期的心电图以及服药后的QT风险类别。作者利用了多逻辑斯特回归模型,输入是所有的风险因素,输出是QTc>450(男)/470(女)的概率,应该是用到了SPSS软件做的,所以作者汇报结果如下:其中第一列是特征名,第二列是权重值,第三列是显著性水平,最后是OR值,方括号里是95%的置信区间。
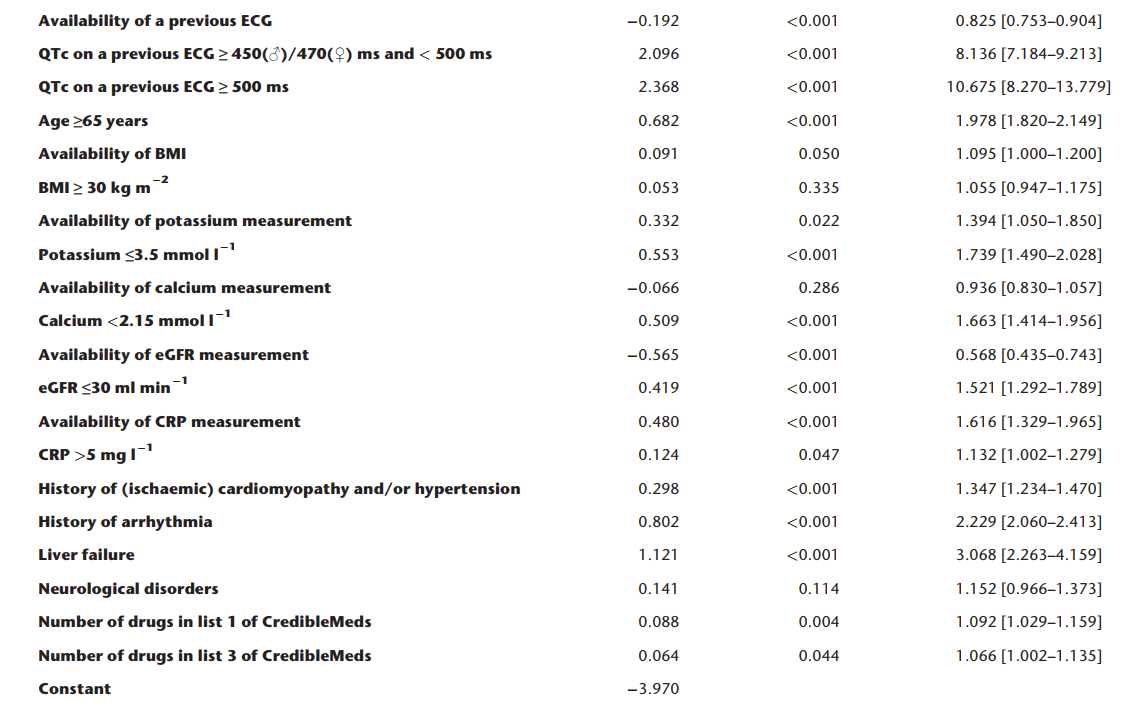
然后就是作者继续分析证明上述结果很好,AUROC达到了0.77左右,然后作者又对患者进行了分群,证明算法在不同分群下效果也还不错。之后作者提出了将这个算法整合到医疗决策系统中,根据模型输出的风险程度会适时给医生进行提醒。
作者在讨论部分提到了整篇文章的优势就是他们有大把的数据,并且是多个医院的。但劣势是数据存在比较多的缺失项,还有就是没有考虑药物之间的相互作用以及病人的遗传特征。
[title]2021年11月7日[/title]
Order Regularization on Ordinal Loss for Head Pose, Age and Gaze Estimation
AAAI 2021 一篇最近做序列回归问题的文章,是阿里巴巴AI中心和三星研究中心联合的工作,文章先回顾了一系列做顺序回归问题的文章,和前几天看到的综述文章稍微不同,这里只提到了在CV方面用的比较多的顺序回归思想,主要就是对不同的类别设定1的个数:有N个类就有N个输出,而其中第Q个类别就应该使得小于Q的所有类别标签为1,比如类别5就应该输出5个1,以此类推,这个思想在单目深度估计,年龄估计问题上都有很多应用。在这种问题里,我们假设模型对于顺序类别的输出应该越来越大。但作者提出这样存在一个问题,如果我们对其中的权重向量不加以约束,那么就会导致决策平面并不是按照我们所想去递增的。具体例子如下:
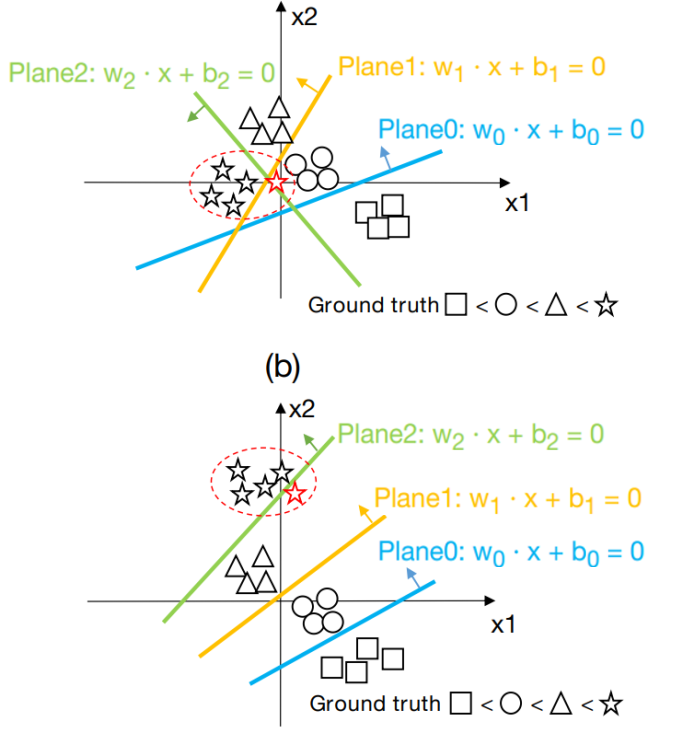
第一张图是未加约束的,这时候三个决策面在黑色的训练数据上做到了随着样本类别增加而值增加,但是当我们来了一个新的样本属于星时,它虽然离星很近,但在模型里会被分成圆形。而用了作者的方法后,各个决策面几乎是平行的,并且满足了顺序关系,这样即使新样本被分错,也不至于太离谱。因此作者提出了3种损失的组合:
Original Ordinal Loss
$\begin{aligned} \operatorname{Loss}_{o}=& \frac{1}{K} \sum_{i=1}^{K} \sum_{n=0}^{N-1}-\left(y_{i}^{n} \log \left(\sigma\left(\mathbf{w}_{n}^{T} \operatorname{Net}\left(S_{i} ; \mathbf{W}_{n e t}\right)+b_{n}\right)\right)\right.\\ &\left.+\left(1-y_{i}^{n}\right) \log \left(1-\sigma\left(\mathbf{w}_{n}^{T} \operatorname{Net}\left(S_{i} ; \mathbf{W}_{n e t}\right)+b_{n}\right)\right)\right) \end{aligned}$
那个小sigma号里面是网络的输出,其中$\mathbf{w}_{n}^{T} $是决策面n的参数,$b_{n}$是决策面的偏移量,前面的求和号是对所有类别在所有样本上的损失求和。
等到了预测阶段,就是数一个样本中>1的label的个数,然后用个数乘以区间长度就是这个样本的最终输出,这一点我一直觉得有点奇怪,其实应该计算的是从最小的类别数开始连续为1的个数,从道理上更合理一些。
Similar-Weights Constraint
这一项约束的是让决策面平行,这里用到了余弦距离。定义权重矩阵(N*C),N为决策面个数,C为特征维数,那么定义矩阵F为两两间的余弦相似矩阵:
$\mathbf{F}=\widetilde{\mathbf{W}} \widetilde{\mathbf{W}}^{T}-\mathbf{I}$
这里一共$\mathbf{N}^{2}-\mathbf{N}$个非零项,那么损失定义为:
$Loss_{\text {plane }}=\left(1-\frac{\sum_{\text {row }=0}^{N-1} \sum_{\text {col }=0}^{N-1} \mathbf{F}_{\text {row,col }}}{N^{2}-N}\right)$
$\quad+\alpha_{\text {var }} \cdot \operatorname{Var}\left(\left\{\left\|\mathbf{w}_{n}\right\|_{2}, n=0,1 \cdots N-1\right\}\right)$
这里作者说第二项的目的是控制权重向量的绝对大小,从而使得网络去寻求改变W的方向来区分样本,而不是靠不同的大小。
Differential-Bias Constraint
差分损失,这部分的目的是为了让每个类别的区间带宽大小控制在一定的范围内,因为权重的方向已经足够相似了,所以控制区间带宽就是控制偏移量$b$,作者利用一个计算技巧定义了这组偏移量:
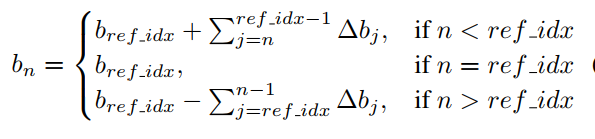
也就是中间的偏移量为自身,而其他类别的偏移量是相应的delta值,举个例子:
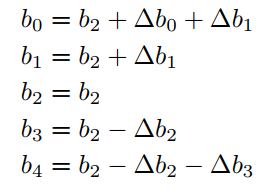
最终的Loss是这三项的总和,然后在一些标准数据集上,作者都达到了SOTA的效果,但个人觉得这里面每部分loss的组合大小是个很tricky的事情,不太清楚调参的效果占多少。
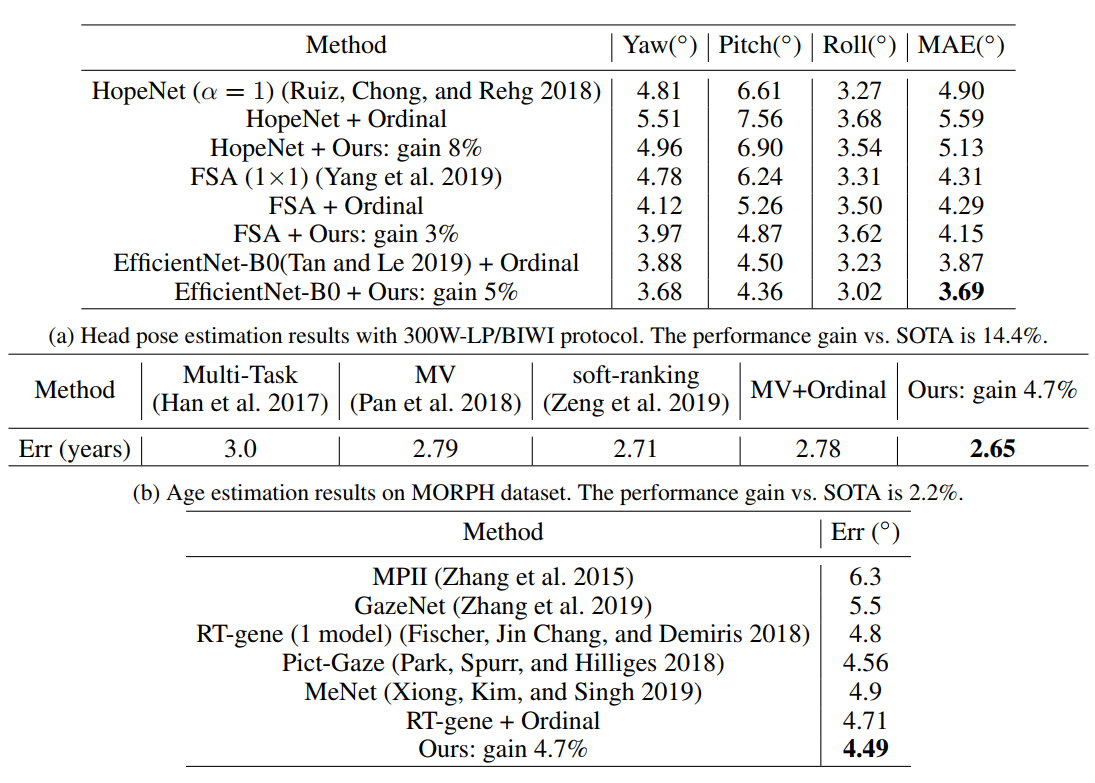
[title]2021年11月8日[/title]
Effects of lead position, cardiac rhythm variation and drug-induced QT prolongation on performance of machine learning methods for ECG processing
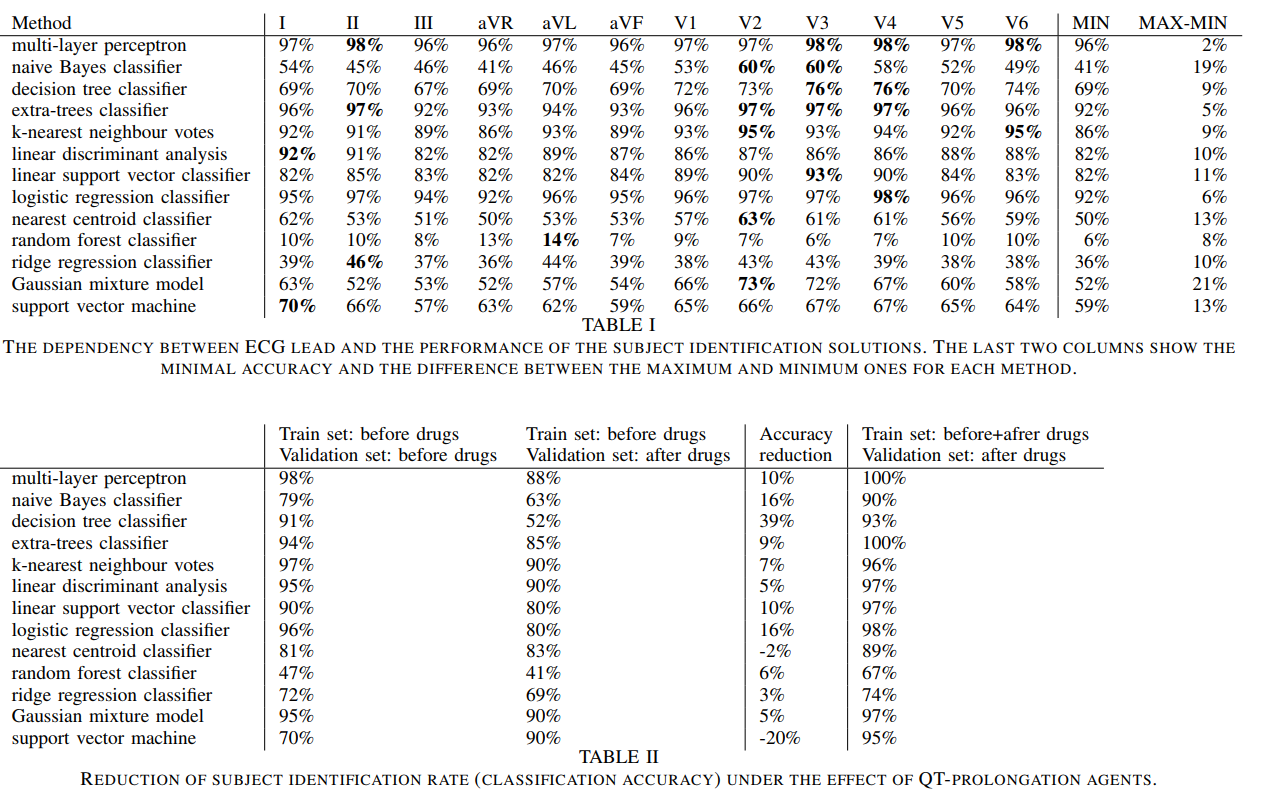
arxiv 2020 作者用了14种算法完成心电图的个体识别任务,关注了其中导联,心电图改变和QT延长对识别结果的影响。结论是,作者发现最影响识别因素的还是算法而不是上面的这些因素,其中MLP和逻辑斯特回归模型的效果最好,但是随机森林的效果很差,甚至比决策树还要差,应该是过拟合了。感觉是个很有意思的现象,看起来如果数据都是同质的,也就是来自于同一个模态,似乎用MLP效果会更好,而如果是表格类数据,那确实应该用决策树家族;但逻辑斯特回归模型在这两类问题上效果好像都还不错,这着实让我有点惊讶。
[title]2021年11月9日[/title]
Managing drug-induced QT prolongation in clinical practice
BMJ 2021 一篇review文章,是一个很好的资源文章。先回顾了QT延长的两种类型:先天型和药物引发的,给出了QT延长的定义和计算方法,提到了QT间期每10ms的增加,会提升5-7%的TdP风险。之后提出了需要考虑的QT延长风险因素,具体有:
1. 性别,年龄大于65,先天因素
2.药物因素,这里提到了药物引发的QT<20ms则考虑为不确定的风险因素,而如果大于20ms则要进行考虑。而联合用药的风险还是不确定的。此外文章中还提到了某些药引发QT延长的区间是已知的。
3.药物相互作用:第一种是药物都是QT风险药物 第二种是A药影响了B药物浓度减少的速率,但是B药会引起QT延长。比如“ritonavir increases the levels of quinidine by decreasing its metabolism (CYP3A4 inhibitor).” 第三种则是药物引起的低钾血症,低镁血症。
之后作者提到了在这个问题上,已经提出了一些风险预测模型,这里提到了5个风险模型,我觉得可以分成3个方面,一方面是简单的评分标准:Tisdale Risk Score for QT Prolongation - MDCalc,另外一个是对于药物互作的评估,还有一个方面是利用之前的信息对患者的延长风险做评估,粗看了一下文章基本上是统计模型。AUC在0.6左右。下面,作者基于以上的这些因素,提出了一种“算法”:
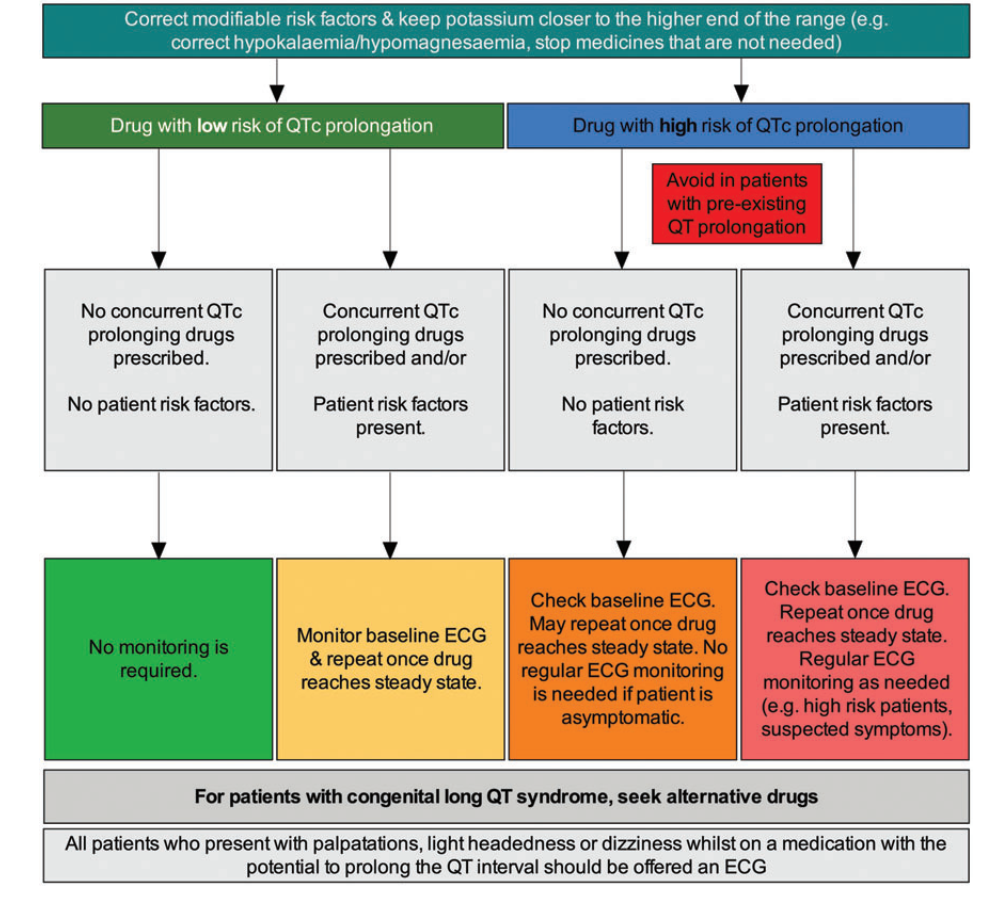
这是类似决策树的一种结构,作者剩下的篇幅都是对这幅图进行了文字描述。作者在最后,提出了接下来关心的几个问题:
- 从QT引发TdP的关键因素是什么?
- 哪些患者服用了延长QT间期的药物,可能从心电图监测中获益?
- 在服用延长QT间期药物的患者中,监测u&e、镁和心电图的安全和理想频率是多少?
[title]2021年11月13日[/title]
Drug-induced QT interval prolongation: mechanisms and clinical management
Therapeutic Advances in Drug Safety 2012 另一篇review文章,在10月份调研到,但没有仔细读。这篇文章主要介绍了hERG通道的微观结构,以及提到了多种能够引发QT延长的药物列表,这个列表应该已经逐渐被QTdrug网站替代了。文章还说QT的检测必须要在质量比较高的数据上进行,这也是现在(2012)年不推荐进行实时QT检测的原因,如今有了深度学习的加持,不知道会不会这个问题有所改观。
此外,文中也提到对药物进行QT延长的实验是药物三期试验中必须要做的一项检测,并且应该能够得到定量的药物延长时间的信息,还需要找到这样的数据。
[title]2021年11月14日[/title]
Cell type ontologies of the Human Cell Atlas
nature cell biology 2021 观点文章 文章主要强调了cell ontology对整个单细胞数据整理偶遇分析的必要性,文中一开始就举了kupffer细胞的例子,他作为免疫细胞,同时还有很多种名字,那么需要一个统一的框架哈编号将多种同义词归为一类,并形成不同概念之间的层次关系,这里细胞之间的相互关系由cell ontology来描绘,而解剖组织结构由UBERON工具来描绘。这两个本体论工具都在单细胞出现之前就已经开始进行了,积累了很丰富的内容,将这两类结合到一起会形成很丰富的内容,如下图所示:

在这个图的右边是多种关系,关系是以属性的形式在kupffer细胞中体现的,换句话说,本身形成的树状结构只是层级关系的一部分,大多数代表了"is a"的关系,而如果我们用part of来建立树的话,应该也会出现层级关系,以及这里的层级只是没有循环图,但是会形成环:
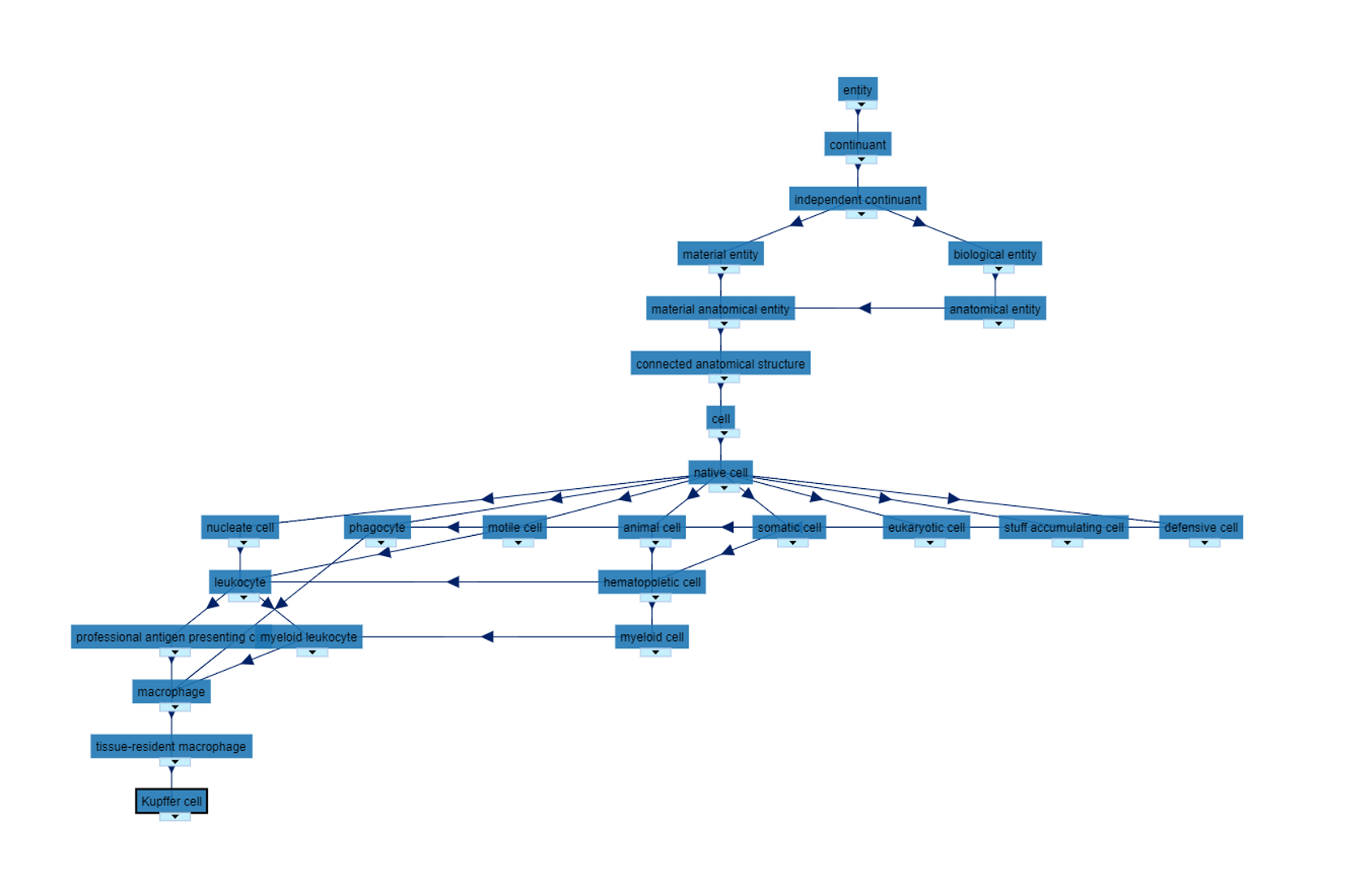
文中也提到了他们的理念是通过多种单细胞数据的分析方法,定义新的细胞类型,确定不同的关系,从而丰富CL树。
[title]2021年11月15日[/title]
Anatomical structures, cell types and biomarkers of the Human Reference Atlas
nature cell biology 2021 观点文章 Human Reference Atlas (HRA) 目的是描绘人体内的所有细胞以供更好的研究。这个工作由16个国际联盟组成,主要是通过三维的解剖结构+anatomical structures, cell types, plus biomarkers (ASCT+B)两部分来推荐,文中举了具体HRA的4个例子。
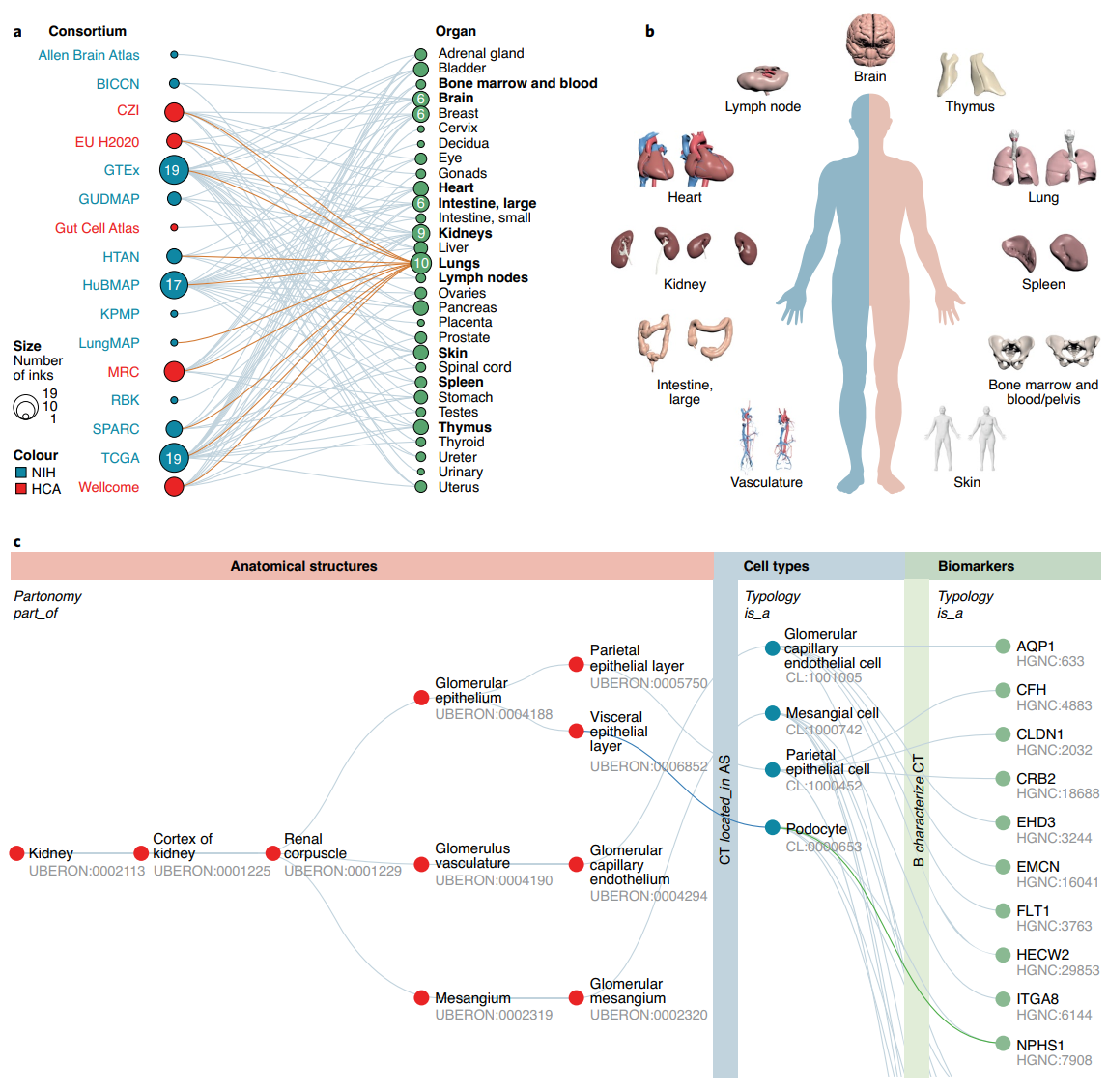
HRA的特色是将空间结构作为组织数据的重要一环,这也和多种空间测序或者空间代谢组学技术的参与有很大的关系。但在处理多种数据时,遇到的最大挑战是没有统一的3D空间参考模型,没有统一的框架和没有统一的语言阻碍了数据的管理,比较和融合。
为了解决这个问题,在2020年3月NIH-HCA召开了50个专家的共同会议,确定了11个器官的三维结构,解剖结构对应的细胞类型,以及对应的生物标记物,这些一共组成了ASCT+B。11个器官有: bone marrow, brain, heart, large intestine, kidneys, lungs, lymph nodes, skin, spleen, thymus, vasculature. 这里的生物标记物是广泛的包含解剖,形态,生理或者组织学上的事物,但目前比较关心的分子标记物。
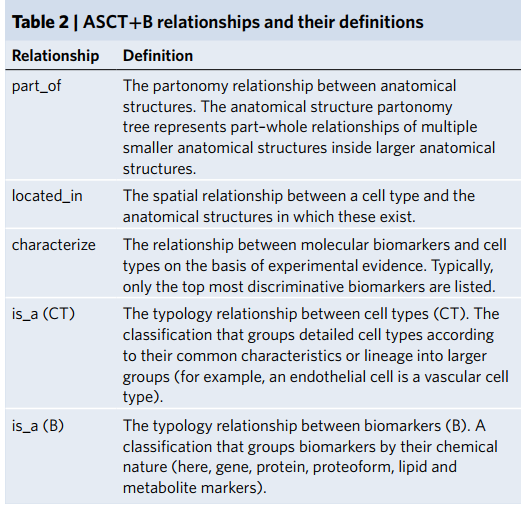
它们是健康组织和细胞身份数据的语义和空间明确的参考,然后可以与疾病设置进行比较。最后,这些表和相关的参考器官可以用来评价细胞类型的语义命名和定义以及它们在解剖学上准确的空间特征方面的进展。
HRA的目标是正确匹配不同人之间的差异性,他们希望所有的结构信息都是一个标准人的,而未来版本的ASCT+B表格可以捕捉和比较捐献者的解剖结构和细胞类型的大小、位置、形状和频率的变化数据。截止到2021年9月,ASCT+B表格已经有11个器官,1424解剖结构,591个细胞类型和1867个生物标记物。这些解剖结构由2543个“part of”关系、4,611个“located_in”关系和3,708个生物标记物和细胞之间的“characterize”关系连接起来,由293个独特的学术出版物和506个网页支持。
作者提出了4种使用ASCT+B的方法:
1.支持实验数据跨组织和尺度的标注和注册
HUBMAP提供了一个可视化的网站,可以在其中查看不同条件(空间,性别)等条件下的数据集,用户可以进行一定的筛选进行数据下载。
2.比较和整合不同组学数据
作者举了一个新冠的例子,我们可以通过选择某个部位上多个组学数据进行分析。
3. 比较健康和疾病数据
在ASCT+B表格中有疾病和健康人的生物标志物,通过比较正常和疾病标记物的不同,我们能得到有关疾病的一些insight。注意这里的数据可能不是单细胞水平的,而是通过三维解剖结构进行映射而得到的。
4. 估计HRA的进度?
如果学术上有新的表格出现,他们的ASCT+B reporter可以比较出新表格从新解剖结构,细胞类型,标志物和实体关系上的贡献,通过记录不同组织对表格的贡献关系,我们可以邀请相关专家一同进行表格的改进。

作者提到工作的局限一方面是对很多解剖结构等结构做了简化,无法完全描绘复杂的人体,另外“健康”的定义将不断演变;用于构建图谱的数据集的HRA元数据包括性别、年龄和种族的信息,以及共病的信息,当纳入标准改变时,可以纳入或排除数据集,并根据需要重新计算HRA。
最初,本体和出版数据,以及器官专家的知识,被编码和统一。随后,将实验数据集与现有的主表进行比较,以确认这些表或根据需要添加到它们中,以捕获健康的人体组织数据。在不久的将来,细胞类型类型将从一个层次扩展到多个层次,还将使用来自Azimuth参考数据,并与细胞本体管理工作密切合作。
随着新的单细胞技术和计算工作流程的发展,解剖结构、细胞类型和健壮的生物标志物的数量可能会增加。因此,表和相关的参考对象是集体工作状态的一个活的“快照”,朝着一个开放的、权威的、可计算的HRA,实验人员可以据此校准他们的数据并作出贡献。HRA的未来用途可能包括跨物种比较或跨物种注释、跨组织/器官比较、健康基因与普通基因或罕见基因的比较
文章评论